呼吸のルーツを探る一酸化窒素還元酵素 (NOR)の構造
〜生体エネルギー変換に関わる膜タンパク質〜
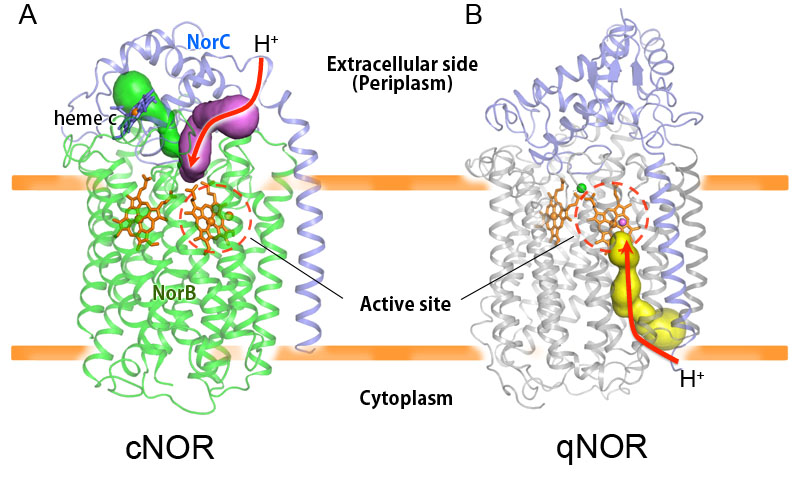
嫌気呼吸で重要な役割を担うNOR
一部の微生物は脱窒反応によって酸素の無い環境でもエネルギーを獲得して生存することができます。これは、酸素の代わりに硝酸塩を電子受容体とする 嫌気的な呼吸です。プロトンが膜外に放出されて得られるプロトン濃度勾配を用いることで生体エネルギーであるATPが合成されています。 その過程で重要な役割を担っているのは、一酸化窒素還元酵素 (NOR)です。NORは脱窒反応を行う細菌や一部の病原菌の細胞膜に存在する金属酵素です。 NORは脱窒の途中で生成してくる有毒なNOを基質として無毒なN2Oに変換しています(NO + 2H+ + 2e- → N2O + H2O)。 NORは、好気呼吸の根幹を担っ ているシトクロム酸化酵素(COX)と約20%のアミノ酸配列の一致があることから、NORとCOXは共通の祖先から進化したと推測されます。つまり、太古の 地球にほとんどO2が無かったことを考えると、NORは呼吸酵素の祖先の形をそのまま残していて、嫌気呼吸から好気呼吸への進化の道筋を解き明かす鍵を 握ると考えられてきました。NORは反応に必要な電子供与体の違いによってcNORとqNORの2種類が知られており、それぞれ2010および2012年にいずれも 当研究室によって初めて立体構造が明らかにされました [1,2]。
cNOR
緑膿菌由来のcNORは、NorC・NorBという2つのサブユニットから構成されます(図1A)。2つのサブユニットの境界には水チャネルがあり、図の上から NORの中心へのプロトンの通り道であると考えられています。cNORの活性部位の非ヘム鉄の配位子となる3つのHis残基は、COXの銅イオンの配位子とし て保存されていますが、その配位構造は異なっており、金属イオンの選択性や基質や反応の違いとなっています。cNORでは非ヘム鉄に配位する残基として Gluが加わっており、この残基がプロトンの供給に関与していると予測されていますが、詳細なメカニズムは未解明なため、現在解析を進めています。
qNOR
一方、キノールを電子供与体とするqNORの立体構造(図1B, Geobacillus stearothermophilus由来のqNOR)では、cNORと比較すると活性部位の構造や膜貫通領域の ヘリックスの配置に多くの類似性があります。Ca2+が2つのヘムbをブリッジしている点や、qNORの親水性ドメインはヘムが無いにもかかわらず シトクロムcフォールドから成っていることも共通点です。しかし、qNORの構造において興味深いのは、細胞質側(図の下側)の分子表面と分子内部を つなぐ水チャネルが存在し、十数個の水分子が極性アミノ酸側鎖とともに水素結合ネットワークを形成して活性部位へとつながっていたことです。 cNORのプロトンの通り道とは反対方向からの水チャネルです。このことは、これまでのNORの生化学的研究を覆す新しい発見となりました。 このqNORの水チャネルの意義を検証するために行った変異体や分子動力学計算に よる解析も、この水チャネルがNOR反応に必要なプロトン伝達経路であることを支持しました。つまり、cNORとは異なり、COXと同一方向の プロトン移動(細胞質→細胞外側)が起こっていることになります。しかも、qNORの水チャネルの位置は、COXで提案されている「Kチャネル」 と呼ばれる経路(細胞質のプロトンが活性部位を経由して細胞外へ汲み出される)と同じ場所にありました。つまり、qNORが持つ水チャネルは、 COXがもつプロトンポンプの「試作品」とも言えるでしょう。
展望
以上のNORの立体構造解析の成果は,生物が酸素を呼吸に利用しはじめた太古の昔に、呼吸酵素がどのようにしてプロトンポンプ機能を 獲得するにいたったのかを詳細に理解するうえで重要な手がかりとなります。サブユニットやドメイン構造や活性部位の構造の特徴は 明らかになりましたが、NORが触媒する反応は未だに理解が進んでいません。我々はX線結晶構造解析や分光解析を用いてNORの触媒反応機構を 詳細に明らかにする研究へと発展させています。[3-6]
[1] T. Hino, Y. Matsumoto, S. Nagano, H. Sugimoto, Y. Fukumori, T. Murata, S. Iwata and Y. Shiro, Science 330, 1666 (2010)
[2] Y. Matsumoto, T. Tosha, A. V. Pisliakov, T. Hino, H. Sugimoto, S. Nagano, Y. Sugita and Y. Shiro, Nature Struct. Mol. Biol. 19, 238 (2012)
[3] N. Sato, S. Ishii, H. Sugimoto, T. Hino, Y. Fukumori, Y. Sako, Y. Shiro, T. Tosha, Proteins 82, 1258-1271 (2014).
[4] T. Tosha, Y. Shiro, IUBMB Life 65, 217-226 (2013).
[5] E. Terasaka, N. Okada, N. Sato, Y. Sako, Y. Shiro, T. Tosha, Biochim. Biophys. Acta, 1837, 1019-26 (2014).
[6] N. Gonska, D. Young, R. Yuki, T. Okamoto, T. Hisano, S. Antonyuk, S. S. Hasnain, K. Muramoto, Y. Shiro, T. Tosha, P. Adelroth, Sci Rep 8, 3637 (2018)
2015年5月執筆(杉本宏), 追記あり